Membres de l'équipe
Irmak Avci
Mariam Dynar
Solène Song
Informatique, Morphogenèse et variabilité
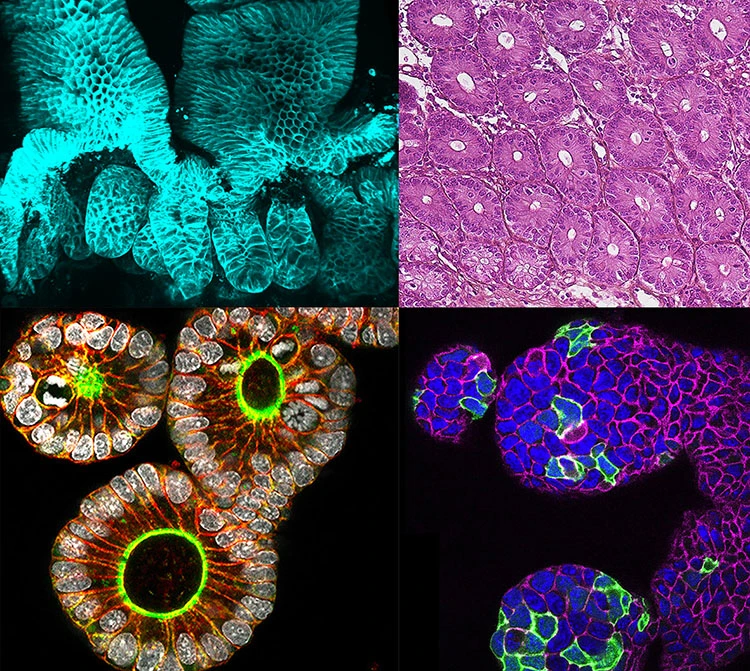
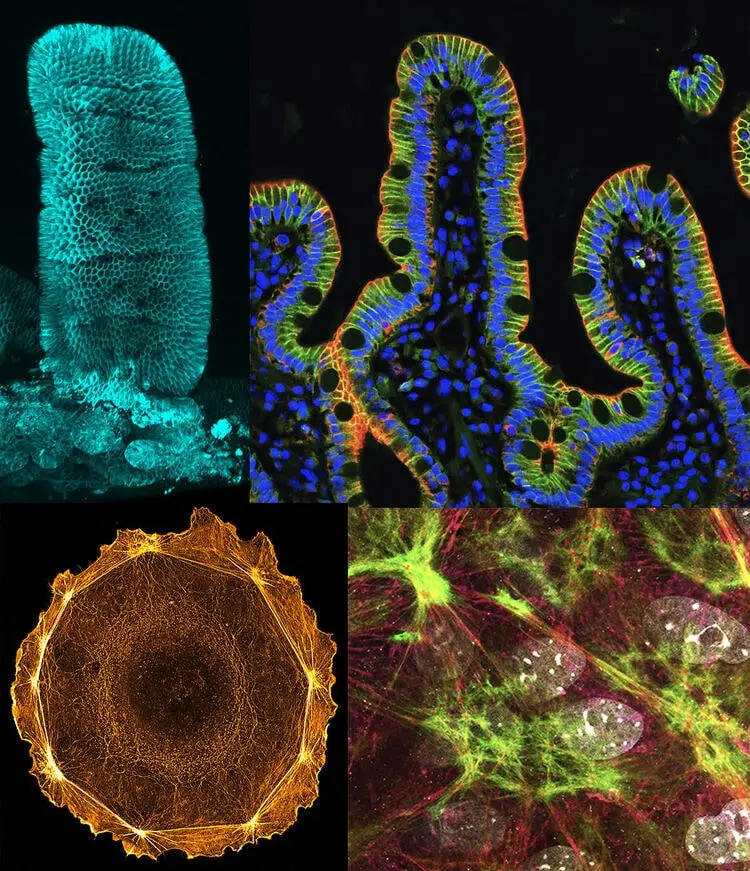
La Théorie des Graphes, l'Apprentissage Automatique et le Big Data animent notre laboratoire et nos analyses de la morphogenèse et de la reproductibilité du développement à travers les espèces à partir d'acquisition par microscopie à fluorescence et spatial omics.
Notre laboratoire se situe à l’interface de l’informatique et de la biologie du développement. Nous nous concentrons sur le développement de méthodes informatiques pour analyser la morphogenèse à l’échelle de la cellule unique dans des organismes entiers tout au long de leur développement.
Notre objectif principal est d’appliquer des algorithmes de vision artificielle, de théorie des graphes, d’apprentissage automatique et de big data pour quantifier et comprendre la reproductibilité du développement au cours de l’embryogenèse dans divers organismes modèles.
Notre but ultime est d’étudier l’impact de la variabilité du développement sur la morphogenèse en utilisant des moyennes statistiques à partir de deux modalités de données principales : les images de microscopie à fluorescence et les omiques spatiales.
Nos recherches s’articulent autour de trois domaines clés :
- Vision par ordinateur : Nous développons des algorithmes de vision par ordinateur et d’apprentissage automatique pour reconstruire et analyser des représentations moyennes d’organismes en développement à l’aide de séries chronologiques d’images de microscopie à fluorescence en 3D. Cela nous permet d’identifier des tendances et des modèles dans les processus de développement.
- Données omiques spatiales : Nous nous intéressons également à l’analyse des graphes, aux structures de données et aux outils de visualisation pour gérer et représenter efficacement des ensembles de données omiques spatiales 3D complexes. Cela permet de découvrir les aspects moléculaires des processus de développement.
- Intégration moyenne des modalités : Notre force principale réside dans l’intégration des images de microscopie à fluorescence et des données omiques spatiales dans des atlas moyens complets de l’embryogenèse à travers de multiples espèces. Ces atlas fournissent des informations précieuses sur les aspects communs et uniques du développement au sein d’une même espèce et d’une espèce à l’autre.
Notre laboratoire entretient des partenariats avec des biologistes du développement et des physiciens à Marseille, Paris (France), Héraklion (Grèce), Cleveland (Ohio, États-Unis), New York (New York, États-Unis) et dans d’autres régions. Grâce à ces collaborations, nous visons à améliorer notre compréhension des processus biologiques fondamentaux tout en promouvant la recherche interdisciplinaire.
Publications
Spatiotemporal transcriptomic maps of whole mouse embryos at the onset of organogenesis
Mathematical and bioinformatic tools for cell tracking
Contact area-dependent cell communication and the morphological invariance of ascidian embryogenesis
MitoMAMMAL: a genome scale model of mammalian mitochondria predicts cardiac and BAT metabolism_edited
Spatiotemporal transcriptomic maps of whole mouse embryos at the onset of organogenesis
Mathematical and bioinformatic tools for cell tracking
Contact area-dependent cell communication and the morphological invariance of ascidian embryogenesis
Actualités
Retour sur les derniers recrutements
Félicitations à Youmna Atieh, Baptiste Libé-Philippot, Léo Guignard, Emmanuelle Leblanc et Séverine Platel.
Nous sommes ravis d’accueillir Leo Guignard et son nouveau groupe de recherche ‘Computer Science, Morphogenesis and Variability’ à l’IBDM.
No jobs opportunities found..