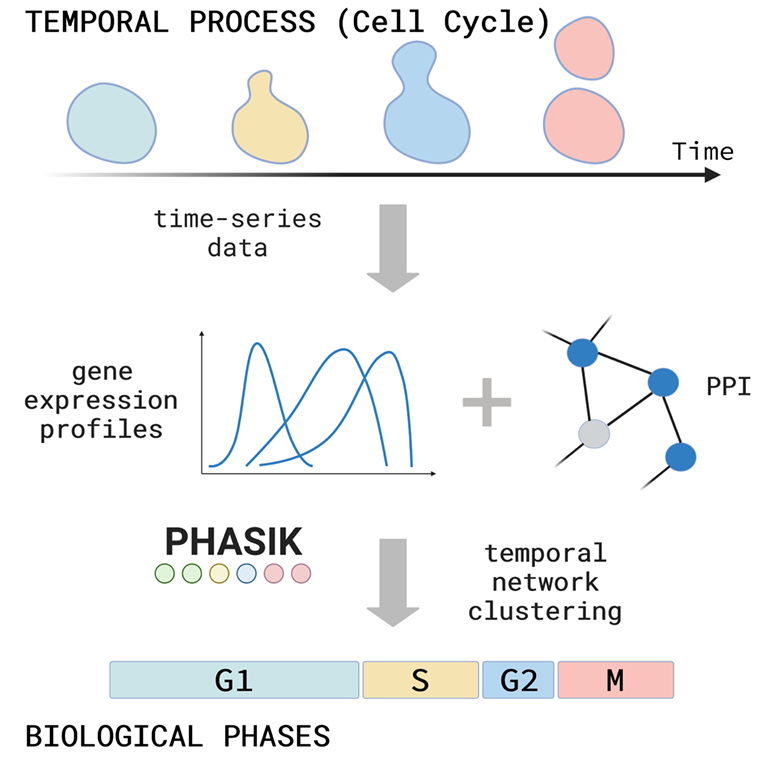
De nombreux systèmes biologiques sont organisés en phases ou états temporels. Le cycle cellulaire, par exemple, comporte 4 phases principales : une première phase d’écart (G1), au cours de laquelle la cellule commence à croître et détermine si elle doit se diviser ou non, suivie d’une phase de synthèse (S) au cours de laquelle l’ADN est répliqué, puis d’une autre phase d’écart (G2), au cours de laquelle la cellule continue à croître et se prépare à la phase finale, la mitose (M), au cours de laquelle le génome dupliqué est séparé en deux cellules filles. Le rythme circadien est un deuxième exemple, dans lequel des gènes clés de l’horloge régulent l’expression des gènes selon un rythme quotidien de 24 heures qui a évolué pour s’adapter aux cycles quotidiens de lumière et d’obscurité. Ces phases ou états temporels d’un système biologique sont créés par la disponibilité, à un moment précis, de protéines régulatrices spécifiques dans une cellule, ainsi que par leurs interactions précises les unes avec les autres Ces changements d’état des cellules ou des systèmes biologiques peuvent être mesurés en quantifiant les niveaux de protéines ou d’ARNm (par approximation) dans des expériences de séries chronologiques.
Nous avons développé une méthode, Phasik, pour extraire les informations de phase de ces séries temporelles en combinant les données d’expression temporelle avec les données d’interaction des réseaux d’interaction de protéines. Nous construisons un réseau (partiellement) temporel, puis nous regroupons les “instantanés” de ce réseau temporel. Nous avons testé Phasik de manière approfondie et avons pu montrer que nous pouvions retrouver de manière fiable les phases et sous-phases connues du cycle cellulaire et détecter l’arrêt du cycle cellulaire chez les mutants. Avec Phasik, nous pouvons également retrouver le rythme de 24 heures de l’horloge circadienne et nous montrons que ce rythme est perturbé chez les mutants du gène de l’horloge.
Phasik est le premier algorithme de regroupement de réseaux temporels appliqué et disponible pour les systèmes biologiques. De plus, c’est la toute première fois que nous démontrons que des informations temporelles partielles pour seulement un sous-ensemble d’interactions dans le réseau sont suffisantes pour extraire les phases d’un système temporel. Phasik peut être utilisé pour déduire les phases d’un large éventail de systèmes biologiques moins connus et peut aider à mieux comprendre leurs mécanismes sous-jacents. Le code est disponible gratuitement (Python, Jupyter).
En savoir plus
Inferring cell cycle phases from a partially temporal network of protein interactions
Authors: Maxime Lucas (IBDM, CPT, I2M), Arthur Morris (University of Oxford), Alex Townsend-Teague (University of Oxford), Laurent Tichit (I2M), Bianca Habermann (IBDM), Alain Barrat (CPT)
Funding: The Turing Centre for Living Systems (CENTURI, ANR-16-CONV-0001)